1que (Italian)
| |||||||
| 1que, resolution 1.80Å () | |||||||
|---|---|---|---|---|---|---|---|
| Ligands: | , | ||||||
| Activity: | Ferredoxin--NADP(+) reductase, with EC number 1.18.1.2 | ||||||
| |||||||
| |||||||
| Resources: | FirstGlance, OCA, RCSB, PDBsum | ||||||
| Coordinates: | save as pdb, mmCIF, xml | ||||||

STRUTTURA AI RAGGI X DELLA FERREDOXIN:NADP+ REDUCTASE DAL CIANOBATTERIO ANABAENA PCC 7119 A 1.8 ANGSTROMSTRUTTURA AI RAGGI X DELLA FERREDOXIN:NADP+ REDUCTASE DAL CIANOBATTERIO ANABAENA PCC 7119 A 1.8 ANGSTROM
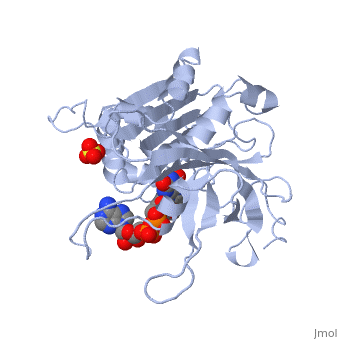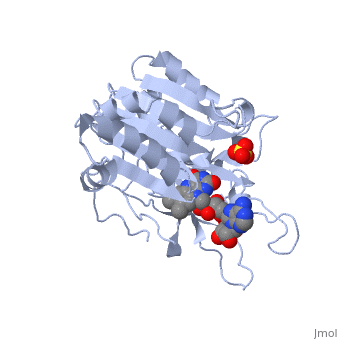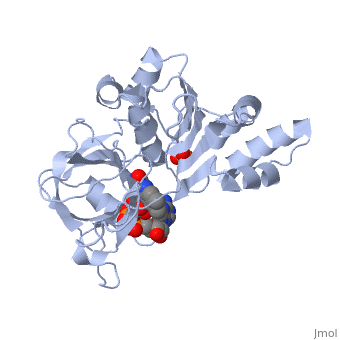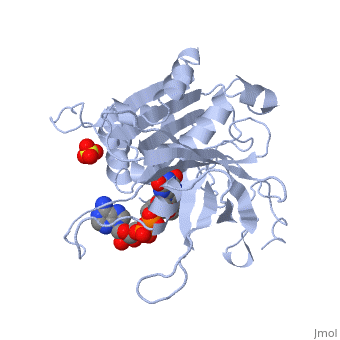
La struttura nel cristallo della ferredoxin:NADP+ reductase (FNR) dal cianobatterio Anabaena PCC 7119 è stata determinata a una risoluzione di 2.6 A con sostituzione isomorfa multipla ed affinata usando dati tra 15.0 a 1.8 A, raccolti a 4 gradi C, fino ad un fattore R di 0.172. Il modello include 303 residui, il cofattore flavin adenine dinucleotide (FAD), un ione solfato localizzato al luogo di legame del NADP+ e 328 molecole di acqua. La struttura di Anabaena FNR, incluso il FAD, una rete di molecole di acqua intrinseche ed una grande cavità idrofobica nel dominio C-terminale, assomiglia a quella dell'enzima di spinacio. Le differenze notevoli concernono l'alfa-elica supplementare corta (residui 172 a 177 in Anabaena FNR), e residui Arg 100 ed Arg 233 i quali legano il NADP+ invece di Lys 116 e Lys 244 nell'enzima di spinacio. Cristalli di un complesso di Anabaena FNR con NADP+ furono ottenuti. Il modello del complesso è stato affinato fino a un fattore R di 0.186, usando dati di diffrazione ai ragi X da 15 fino a 2.25 A di risoluzione raccolti a -170 gradi C. Questo modello include 295 residui, il FAD, il NADP+ intero (con un'occupazione di 0.8) e 444 molecole di acqua. La parte 2'-5' adenine del NADP+ si lega alla proteina come il 2'-fosfo-5'-AMP alla FNR di spinacio. La parte nicotinamide è girata verso la superficie della proteina invece di accatastarsi sopra l'annello isoalloxazine del FNR, come sarebbe richiesto per il trasferimento di idride. Il modello del complesso è in accordo con studi biochimichi precedenti, visto che i residui Arg 100 ed Arg 233 sono implicati nel collegamento del NADP+, e i residui Arg77, Lys 53 e Lys 294 situati sul lato FAD dell'enzima, rimangono liberi per interagire con ferredossina e flavodossina, i partner fisiologici della ferredoxin:NADP reductase.
X-ray structure of the ferredoxin:NADP+ reductase from the cyanobacterium Anabaena PCC 7119 at 1.8 A resolution, and crystallographic studies of NADP+ binding at 2.25 A resolution., Serre L, Vellieux FM, Medina M, Gomez-Moreno C, Fontecilla-Camps JC, Frey M, J Mol Biol. 1996 Oct 18;263(1):20-39. PMID:8890910
From MEDLINE®/PubMed®, a database of the U.S. National Library of Medicine.
Riassunto tradotto da MEDLINE®/PubMed®, una banca dati del U.S. National Library of Medicine.
A proposito di questa StrutturaA proposito di questa Struttura
1QUE è una struttura a 1 catena di sequenza di Anabaena sp.. Dalle informazioni cristallografiche completi sono disponibili su OCA.
RiferimentoRiferimento
- ↑ Serre L, Vellieux FM, Medina M, Gomez-Moreno C, Fontecilla-Camps JC, Frey M. X-ray structure of the ferredoxin:NADP+ reductase from the cyanobacterium Anabaena PCC 7119 at 1.8 A resolution, and crystallographic studies of NADP+ binding at 2.25 A resolution. J Mol Biol. 1996 Oct 18;263(1):20-39. PMID:8890910 doi:10.1006/jmbi.1996.0553
Page seeded by OCA on Tue Feb 17 04:01:53 2009